Developmental Cell | 李栋课题组揭示MALAT1-m6A修饰的生物学功能
N6-甲基腺苷(m6A)修饰是一种普遍存在且有重要生物学功能的RNA化学修饰。细胞内几乎所有类型的RNA分子中均广泛存在m6A修饰。RNA分子m6A修饰状态的动态变化及其功能主要由m6A甲基转移酶(Writer)、m6A去甲基化酶(Eraser)和m6A阅读蛋白(Reader,即“读码器”)等蛋白复合物共同调控。目前,大多数研究均通过调控m6A Writer,Eraser或Reader蛋白的活性来研究m6A修饰的生物学功能。然而,需要注意的是上述研究策略改变了整个转录组的m6A状态,最终表型取决于来自不同类型转录本m6A状态变化的综合效应,导致难以确定究竟哪种或者哪类转录本的m6A修饰对最终表型起主要作用,甚至导致相互矛盾的结论,例如:特异性抑制m6A去甲基化酶FTO(即RNA的m6A总体水平上调)抑制胶质母细胞瘤的进展【1】;但与这一结论相反,沉默m6A甲基化酶METTL3(即RNA的m6A总体水平下降)也可抑制胶质母细胞瘤的进展【2】。因此,尤为需要开展具有转录本特异性的m6A功能研究。MALAT1基因在脊椎动物中高度保守,其转录本为一种长度约为8 kb的长链非编码RNA(long noncoding RNA, lncRNA),在核斑上高度富集。许多研究已发现lncRNA MALAT1的高表达与非小细胞肺癌、食管癌等多类肿瘤的侵袭转移风险高度正相关【3,4】。但是,MALAT1如何促进侵袭转移发生的分子机制一直尚未明确。
2021年2月20日,中国科学院生物物理研究所李栋课题组在Developmental Cell杂志以长文(Article)形式发表题为 “N6-methyladenosine modification of MALAT1 promotes metastasis via reshaping nuclear speckles” 的论文。该研究综合运用长时程超分辨率活细胞成像、CUT&Tag测序和定量蛋白质组学等技术方法,揭示了:lncRNA MALAT1存在m6A修饰富集区,且MALAT1-m6A对维持核斑(Nuclear Speckles)组分动态平衡,进而调控食管癌细胞的增殖和侵袭转移能力起关键作用。然而,通过传统方式调控m6A Writer(METTL3)或Reader(YTHDC1)并不能复现上述表型,提示了研究转录本特异性m6A修饰的重要性和必要性。

作者对食管癌细胞系KYSE-30进行meRIP-seq,并比对RMBase数据库中多个独立的测序数据发现MALAT1富含30个高度可信的m6A修饰位点,其中的28个集中分布在长度约为1.5 kb且高度保守的区域内(chr11:65,499,435-65,500,994)。基于此,作者构建了特异性删除MALAT1基因中富集m6Amotif区域(MALAT1-∆m6A)的KYSE-30和KYSE-150细胞系。RNA-seq结果表明虽然MALAT1基因表达量在缺失m6A修饰后几乎没有变化,但数百个癌症相关基因的表达量在MALAT1-∆m6A细胞中却发生了显著变化,并展现出增殖和侵袭转移能力显著抑制的表型。而且,在MALAT1-∆m6A细胞中回补表达野生型MALAT1能较大程度恢复其受损的增殖和侵袭转移能力,但表达m6A突变型MALAT1却不能。另一方面,沉默m6A Writer(METTL3)或Reader(YTHDC1)所引发的基因表达变化主要影响RNA加工等过程,对细胞的增殖和侵袭能力无显著影响。这些结果表明: (1)特异性改变单个转录本的m6A修饰与改变转录组整体m6A修饰产生的表型大相径庭;(2) 调控单个lncRNA MALAT1的m6A修饰即可引起癌症相关基因表达谱的整体变化。

图1 . 野生型和MALAT1-∆m6A细胞中RBM15斑点与核斑互作,以及YHDC1在核斑中富集程度的对比。
为阐明MALAT1-m6A如何调控侵袭转移,作者首先利用RNA免疫沉淀证明了MALAT1-m6A修饰是由甲基转移酶复合物METTL3/WTAP/RBM15催化形成,并被YTHDC1蛋白阅读识别;其次,利用定量蛋白质谱发现MALAT1-∆m6A能够导致几十种核斑定位蛋白在核斑上富集程度降低;最后,利用自主开发的3D-SIM超分辨显微镜和光片显微镜系统【5,6】对比了野生型和MALAT1-∆m6A细胞中RBM15、YTHDC1、SF3A1、PABPN1与核斑的共定位、动态互作等性质的差别。成像结果表明:与野生型细胞相比,在MALAT1-∆m6A细胞中,RBM15斑点与核斑的互作(contact)显著减少,同时YTHDC1、SF3A1和PABPN1在核斑中的富集显著降低;而且这些蛋白在核斑富集水平的降低可以被野生型MALAT1恢复,但无法被m6A突变型MALAT1恢复。超分辨成像结果与生化和质谱的实验结论相互印证,共同揭示 lncRNA MALAT1的m6A修饰对维持核斑组分稳态起重要作用。
核斑组分的变化又如何导致癌症相关基因表达的改变?最近的研究表明核斑附近为转录活性区域【7】,基因座位靠近核斑通常可增强该基因的表达【8】。基于此,作者推测MALAT1-∆m6A引起的核斑组分变化可能改变核斑与相关基因座位的结合程度,进而改变其转录水平。作者利用CUT&Tag测序发现核斑与相关基因的结合位点主要位于这些基因的转录起始位点,而MALAT1-∆m6A显著减弱了核斑与转录起始位点的结合,这其中包括 420个在MALAT1-∆m6A细胞中转录下调的基因。作者进一步选取JUN和TWIST2两个代表性原癌基因,用3D-SIM超分辨成像统计了免疫荧光标记的核斑与DNA-FISH标记的基因座位的相对距离,结果表明MALAT1-∆m6A使上述两个基因与核斑的接触频率显著下降,这印证了CUT&Tag测序结果。超分辨成像与CUT&Tag测序结果共同证明MALAT1的m6A修饰能够调控核斑与基因组中特定座位的结合,进而调控相关基因的表达。
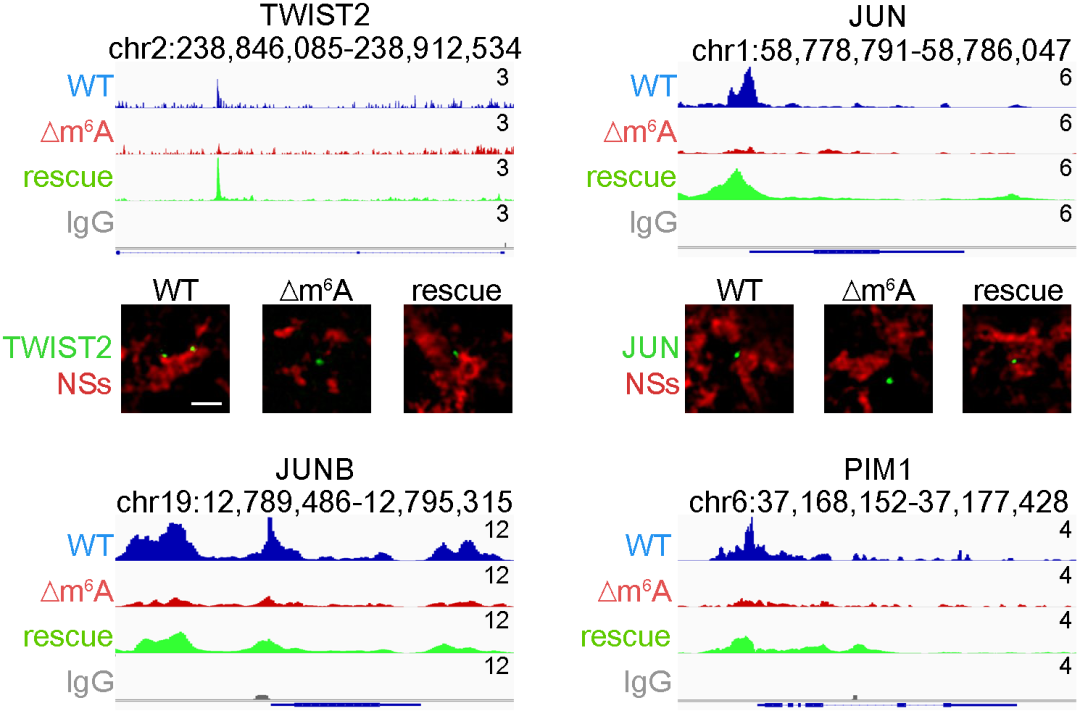
图2. CUT&Tag测序和3D-SIM超分辨成像揭示核斑与4个代表性原癌基因的结合程度在野生型,MALAT1-∆m6A,以及特异性回补YTHDC1细胞系中的变化。
作者进一步在m6A缺陷的MALAT1基因座位敲入MS2适配子序列,以此实现m6A reader蛋白YTHDC1的特异性回补(MALAT1-∆m6A+MCP-YTHDC1)。结果显示,通过在lncRNA MALAT1-∆m6A上特异性结合YTHDC1可重新富集大多数含量下调的核斑蛋白,进而恢复核斑与癌症相关基因的结合,促进其表达,最终显著恢复MALAT1-∆m6A细胞的增殖和侵袭转移能力。最后,作者通过尾静脉分别注射野生型、MALAT1-∆m6A,和MALAT1-∆m6A+MCP-YTHDC1细胞系到免疫缺陷小鼠,结果表明:相比野生型细胞,MALAT1-∆m6A细胞显著降低了肺部转移病灶的发生,而MALAT1-∆m6A+MCP-YTHDC1细胞则明显恢复了肺部转移能力,验证了上述结论。

图3. MALAT1通过其m6A修饰调控基因表达示意图
综上,本研究展示了超分辨显微成像提供的时空动态信息与分子生化、质谱、测序等实验结果互为补充和印证,从多角度揭示了lncRNA MALAT1的m6A修饰在癌细胞增殖和侵袭转移能力中的关键作用。
中科院生物物理研究所李栋课题组助理研究员王新禹博士(兼共通讯作者)、博士研究生刘冲、张思微为该论文的共同第一作者;中科院生物物理研究所卜鹏程课题组对动物实验给予了大力帮助;中国科学院生物物理研究所、广州生物岛实验室李栋研究员为该论文的通讯作者(Lead contact)。本研究得到了科技部、国家自然科学基金委、中国科学院、腾讯“科学探索奖”的资助。
注:文章引用《BioArt》:https://mp.weixin.qq.com/s/4GZ-1rse8UTgiZ7nTruYlQ
原文链接: DOI: https://doi.org/10.1016/j.devcel.2021.01.015
参考文献:
1. Cui, Q., et al., m(6)A RNA Methylation Regulates the Self-Renewal and Tumorigenesis of Glioblastoma Stem Cells. Cell Reports, 2017. 18(11): p. 2622-2634.
2. Visvanathan, A., et al., Essential role of METTL3-mediated m(6)A modification in glioma stem-like cells maintenance and radioresistance. Oncogene, 2018. 37(4): p. 522-533.
3. Ji, P., et al., MALAT-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer. Oncogene, 2003. 22(39): p. 8031-41.
4. Wang, X.Y., et al., Silencing of long noncoding RNA MALAT1 by miR-101 and miR-217 Inhibits Proliferation, Migration, and Invasion of Esophageal Squamous Cell Carcinoma Cells. Journal of Biological Chemistry, 2015. 29(7): p. 3925-3935.
5. Guo, Y.T., et al., Visualizing Intracellular Organelle and Cytoskeletal Interactions at Nanoscale Resolution on Millisecond Timescales. Cell, 2018. 175(5): p. 1430.
6. Li, D., et al., ADVANCED IMAGING. Extended-resolution structured illumination imaging of endocytic and cytoskeletal dynamics. Science, 2015. 349(6251): p. aab3500.
7. Quinodoz, S.A., et al., Higher-Order Inter-chromosomal Hubs Shape 3D Genome Organization in the Nucleus. Cell, 2018. 174(3): p. 744-757 e24.
8. Kim, J., et al., Gene expression amplification by nuclear speckle association. J Cell Biol, 2020. 219(1).
微信公众号
Visualize your ideas
广州市番禺区大学城中心北大街66号创新大厦110-112
安徽省合肥市高新区合欢路16号天怡新世纪商务中心研发生产楼西楼二层


